-
Notifications
You must be signed in to change notification settings - Fork 30
Commit
This commit does not belong to any branch on this repository, and may belong to a fork outside of the repository.
- Loading branch information
Showing
1 changed file
with
138 additions
and
0 deletions.
There are no files selected for viewing
This file contains bidirectional Unicode text that may be interpreted or compiled differently than what appears below. To review, open the file in an editor that reveals hidden Unicode characters.
Learn more about bidirectional Unicode characters
| Original file line number | Diff line number | Diff line change |
|---|---|---|
| @@ -0,0 +1,138 @@ | ||
| # 生信爱好者周刊(第 156 期):说说2024你的收获吧~ | ||
|
|
||
| 这里记录每周值得分享的生信相关内容,周日发布。 | ||
|
|
||
| 本杂志开源(GitHub: [openbiox/weekly](https://github.com/openbiox/weekly "openbiox/weekly")),欢迎提交 issue,投稿或推荐生信相关内容。 | ||
|
|
||
| [「生信周刊讨论区」](https://github.com/openbiox/weekly/discussions "「生信周刊讨论区」") | ||
|
|
||
| ## 封面图 | ||
|
|
||
|  | ||
| [GPU paints the Mona Lisa](https://codingstuff.substack.com/p/if-gpus-are-so-good-why-do-we-still "GPU paints the Mona Lisa") | ||
|
|
||
| ## 本周话题:说说2024你的收获吧~ | ||
|
|
||
| @kkjtmac 简短概括一下:2024在科研、技术开发和项目管理方面取得了一定的进展,更加深入探索细胞与基因治疗等领域,推动多个技术平台应用。 | ||
|
|
||
| ## 生信研究 | ||
| 1、[Nature Genetics | 一种高通量、大视野的空间转录组学新技术(MAGIC-seq)](https://mp.weixin.qq.com/s/eRMzPe-EdUfvVFfmrCU6nQ) | ||
|
|
||
| 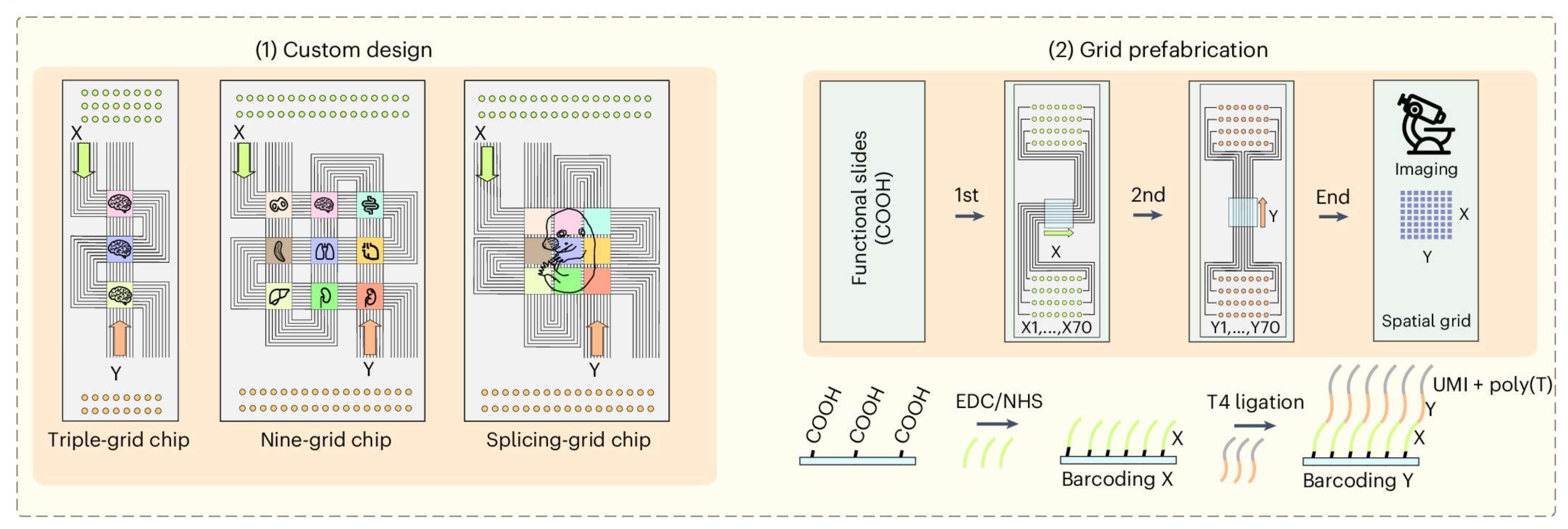 | ||
|
|
||
| Nature Genetics 上这篇文章报道了中国科学院动物研究所赵方庆团队在空间转录组学领域取得的重大突破。他们开发了一种名为 MAGIC-seq 的高通量、大视野空间转录组学新技术,通过创新的网格化微流控芯片设计和新的空间编码技术,显著提高了检测通量和捕获面积,同时降低了成本和批次效应。该技术首次提出了“拼接芯片”的概念,能够灵活调整网格布局,适应不同样本形状和数量的需求。MAGIC-seq在小鼠多个组织(包括大脑、小脑、心脏等)的测试中表现出色,成功构建了高质量的三维空间转录组图谱,为空间转录组学研究提供了强大的工具。 | ||
|
|
||
| - 论文链接:https://www.nature.com/articles/s41588-024-01906-4 | ||
|
|
||
| 2、[Nature | AI从头设计蛋白,攻克百年难题](https://mp.weixin.qq.com/s/r8YnPlBA4S-VpNdRItr8ew) | ||
|
|
||
| 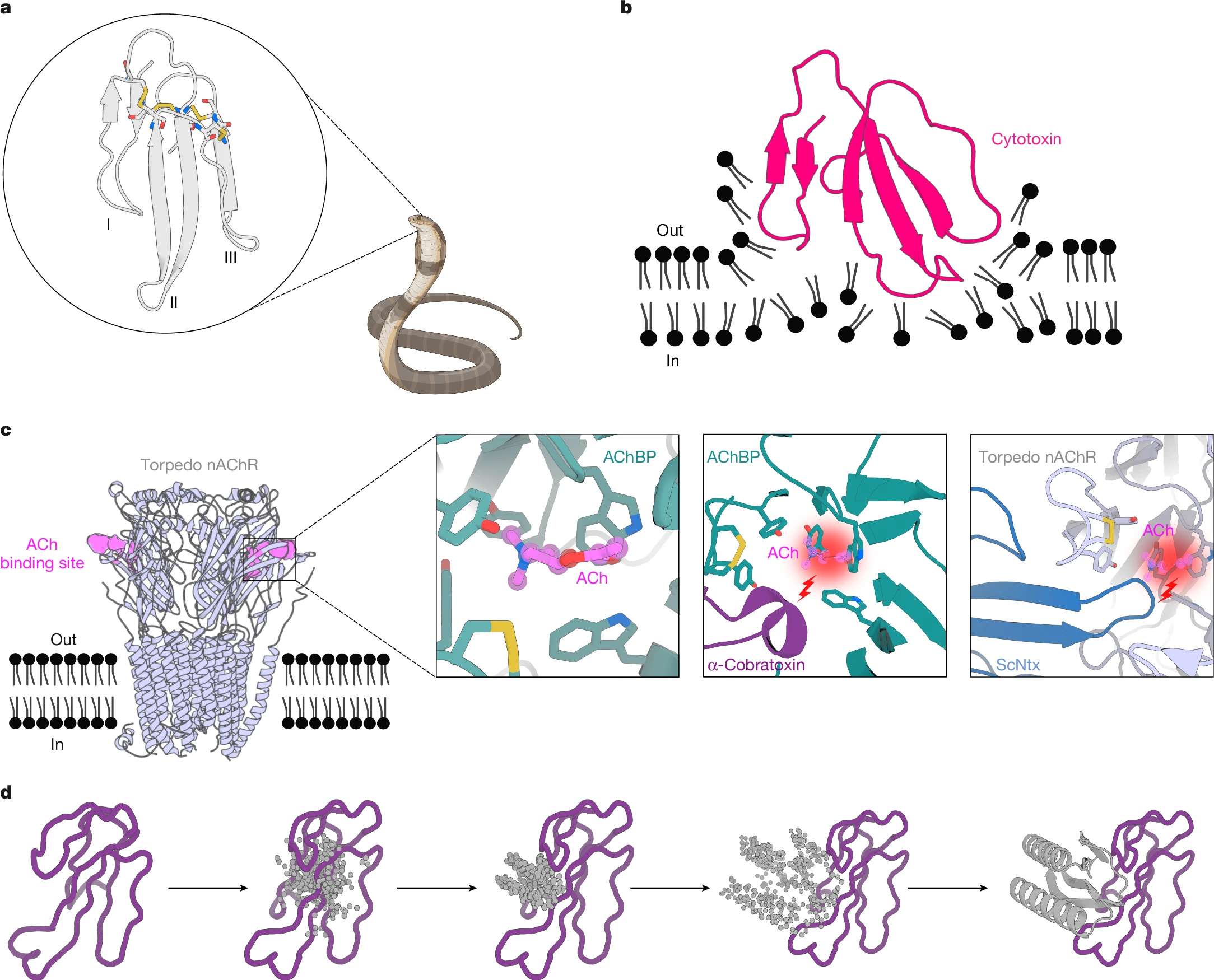 | ||
|
|
||
| 诺贝尔化学奖得主David Baker教授团队在《Nature》发表成果,利用AI从头设计的蛋白质成功阻断了眼镜蛇、蝰蛇等毒蛇毒液中致命毒素的作用。研究团队通过AI工具RFdiffusion设计出“迷你结合剂”,能够紧密结合蛇毒中的关键毒素成分。实验表明,这些结合剂在体外和体内均能有效中和毒素,甚至在小鼠被注射致死剂量毒素15分钟后,仍能实现完全保护。该研究不仅为蛇咬伤治疗提供了新的思路,还展示了AI在蛋白质设计领域的巨大潜力。 | ||
|
|
||
| - 论文链接:https://www.nature.com/articles/s41586-024-08393-x | ||
|
|
||
| 3、[Genome Biology | 表观基因组学预训练语言模型EpiGePT](https://mp.weixin.qq.com/s/1yWV6D2fzrZhrCYs0RQGxg) | ||
|
|
||
| 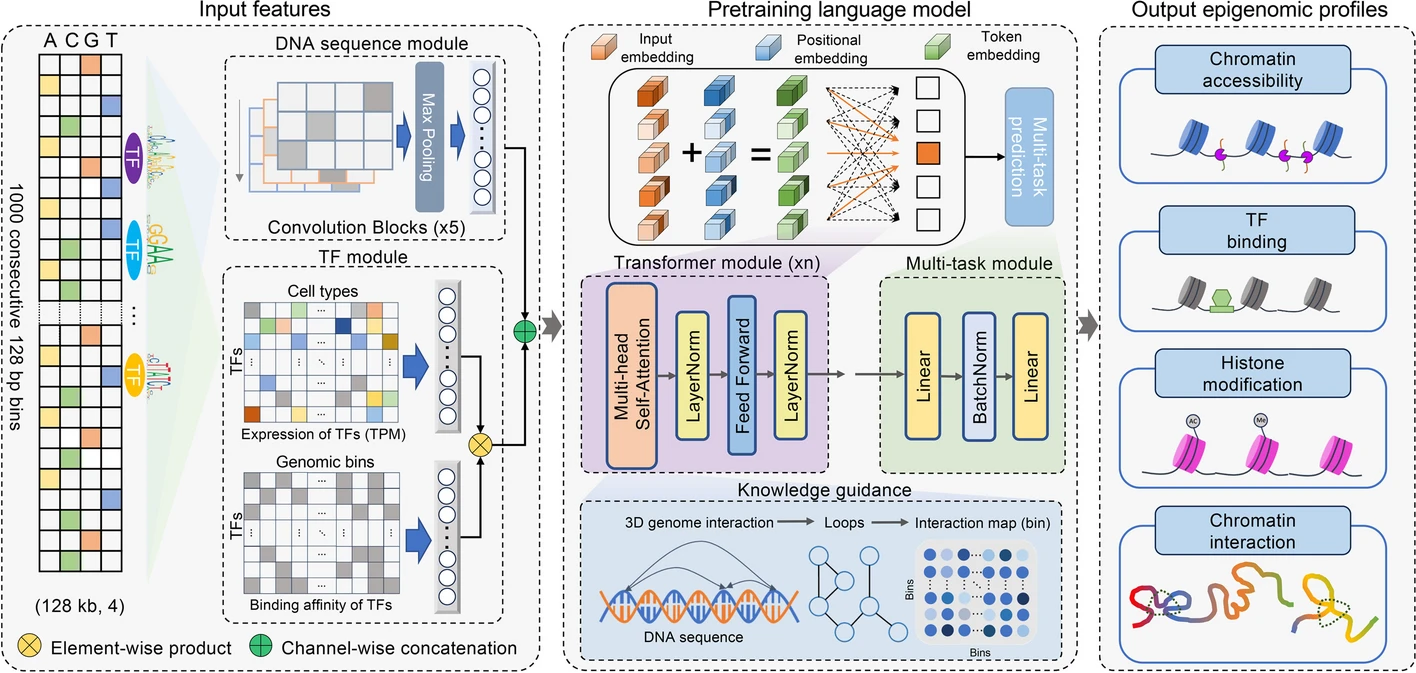 | ||
|
|
||
|
|
||
| 人类基因组中非编码区域的调控机制一直是基因组学的难题。传统基因组语言模型在预测新细胞类型的表观基因组信号方面存在局限。Genome Biology上发表提出了EpiGePT模型。该模型基于Transformer架构,融合DNA序列、转录因子表达和结合基序信息,突破了传统模型的局限,能够准确预测染色质开放性、组蛋白修饰等多种表观基因组信号,并具备细胞类型感知能力。EpiGePT通过三维基因组数据引导训练,增强了对染色质互作关系的捕捉能力,验证了其在人类和小鼠细胞系中的高准确性和泛化能力。该模型为解码基因调控机制、解读致病变异提供了有力支持,并有望推动精准医学和靶向药物研发。 | ||
|
|
||
| - 论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03449-7 | ||
|
|
||
| 4、[Nature Methods | 核苷酸转换模型(Nucleotide Transformer)](https://mp.weixin.qq.com/s/L203QY9eQyDV0rd7A1FrMQ) | ||
|
|
||
| 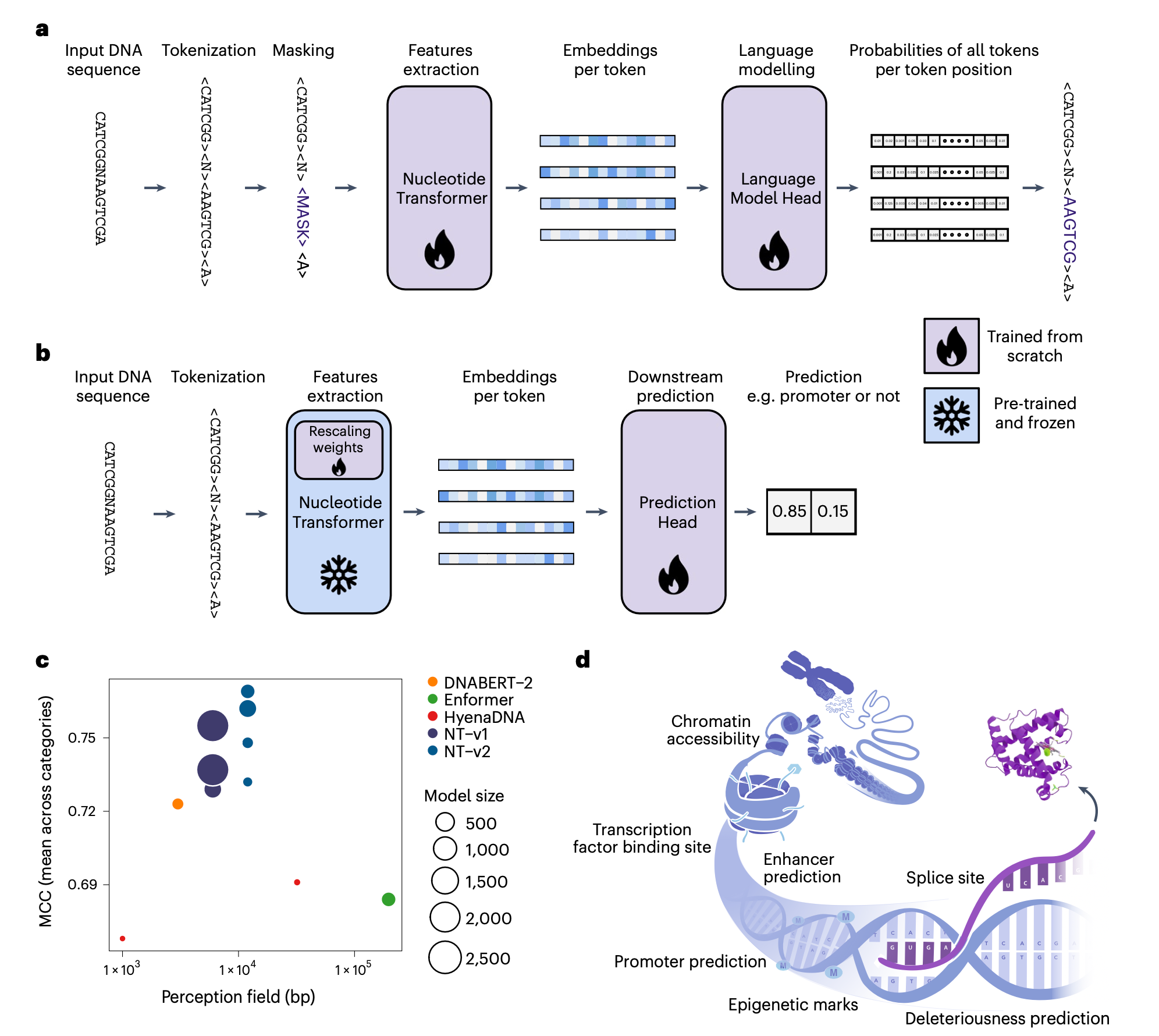 | ||
|
|
||
| 近几年,人工智能(AI)领域的基础模型为这一难题带来了新曙光。这些模型(如BERT、GPT)通过从文本中学习语言规则,在自然语言处理中实现了从无到有的认知飞跃。受此启发,研究人员将这一技术嫁接到基因组学中,开发出了专门用于DNA序列分析的核苷酸转换模型(Nucleotide Transformer, NT)。它不仅能像阅读文本一样解读DNA序列,还能够预测基因组中的关键功能区域。更令人兴奋的是,NT模型通过跨物种的数据训练,展现了强大的任务适应性和功能泛化能力。无论是剪接位点的精确定位,还是复杂增强子活性的预测,这一模型都打破了传统方法的桎梏,为基因组学研究提供了前所未有的洞见。 | ||
|
|
||
| - 论文链接: https://doi.org/10.1038/s41592-024-02523-z | ||
|
|
||
| ## 博文资讯 | ||
|
|
||
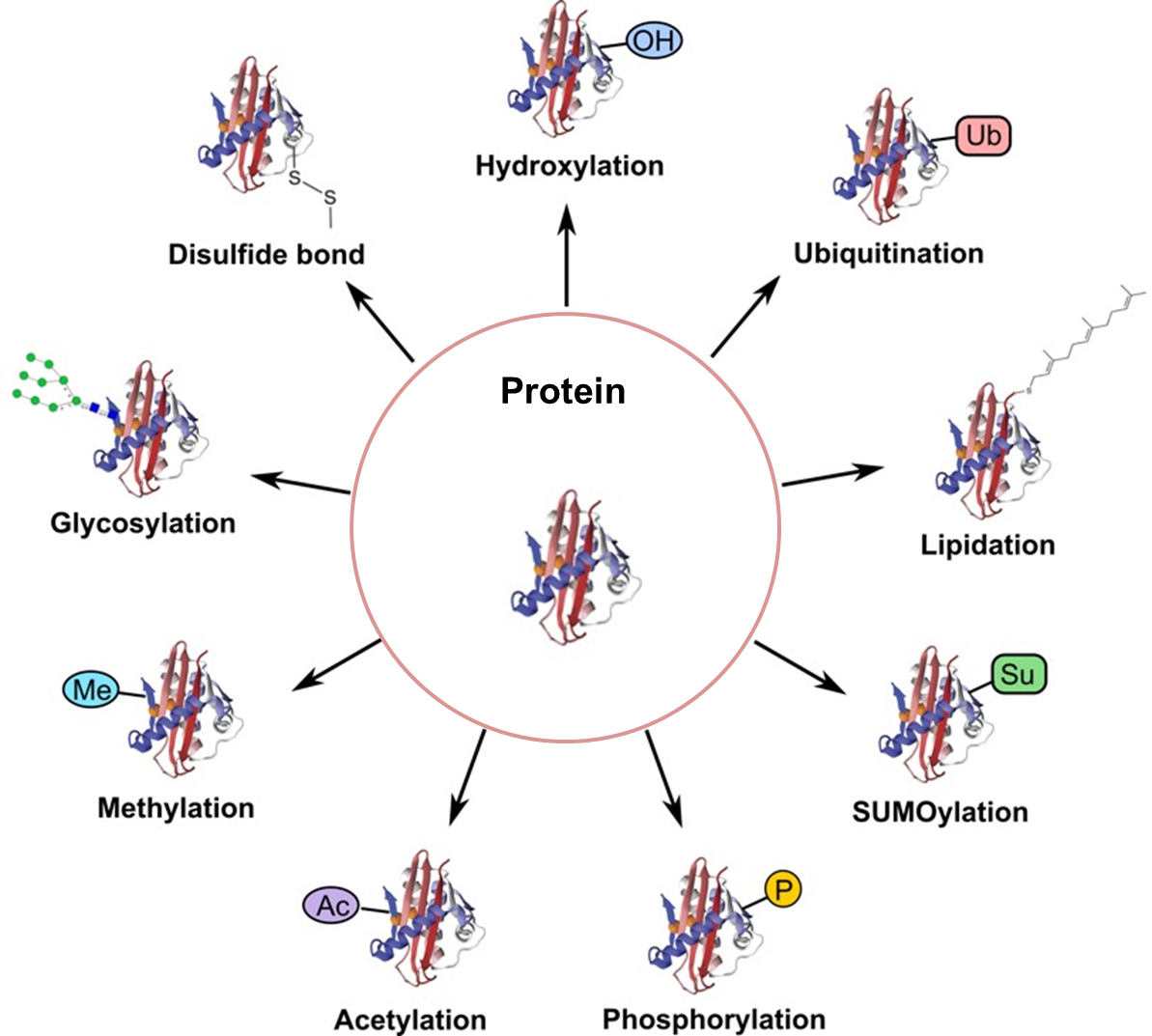
| 5、[蛋白翻译后修饰:从1906到2024](https://mp.weixin.qq.com/s/K1ZZCUDgFXGa_-9ZDHRxSw) | ||
|
|
||
|  | ||
|
|
||
| 蛋白翻译后修饰(PTMs)是指蛋白质在生物合成后氨基酸侧链的化学修饰,目前已发现超过400种PTMs类型,对蛋白质功能有深远影响。文章回顾了PTMs的历史发展,介绍了经典和新兴的PTMs类型。经典PTMs包括磷酸化(1906年首次发现,调控蛋白质活性,参与细胞信号传导等过程)、乙酰化(1964年发现,影响染色质稳定性和蛋白质相互作用)和泛素化(1975年发现,调控蛋白质降解和信号转导)。新兴PTMs包括巴豆酰化(2011年发现,标记活跃启动子和增强子)和乳酸化(2019年发现,参与基因表达和代谢调节)。这些修饰的研究不断拓展,为理解蛋白质功能和疾病机制提供了重要视角。 | ||
|
|
||
| 6、[2025年度癌症报告](https://mp.weixin.qq.com/s/aQ20OfKEAdKbdxfBpVwA9A) | ||
|
|
||
|  | ||
|
|
||
| 2025年1月16日,《临床医师癌症杂志》发布了《2025年度癌症报告》。报告指出,从1991年到2022年,美国癌症死亡率下降了34%,避免了约450万人死亡。2025年预计美国将有2,041,910例新癌症诊断(每天5,600例),618,120例癌症死亡病例。前列腺癌、肺癌和结直肠癌占男性发病的48%,乳腺癌、肺癌和结直肠癌占女性发病的51%。尽管吸烟率从1965年的42%下降到2020年的12%,肺癌仍是主要死亡原因。癌症发病率在中年群体中增加,尤其是女性。儿童和青少年癌症发病率略有下降,但死亡率显著降低。报告强调,癌症负担正在从老年人向年轻人、从男性向女性转移,乳腺癌、前列腺癌和子宫癌发病率上升需关注。 | ||
|
|
||
| 7、[如果 GPU 那么好,为什么我们还用 CPU](https://codingstuff.substack.com/p/if-gpus-are-so-good-why-do-we-still "如果 GPU 那么好,为什么我们还用 CPU") | ||
|
|
||
|  | ||
|
|
||
| 这篇文章探讨了尽管GPU(图形处理单元)在处理并行计算任务方面表现出色,但CPU(中央处理单元)仍然被广泛使用的原因。文章指出,CPU在处理复杂逻辑、顺序执行以及低功耗场景中具有独特优势。例如,CPU更适合运行操作系统、处理多任务和执行复杂的算法,而GPU则在图形渲染、深度学习等并行任务中表现更好。此外,CPU的通用性和兼容性使其能够适应各种应用场景,而GPU则需要特定的硬件和软件支持。文章强调,CPU和GPU各有优势,未来可能会继续共存,而不是一方完全取代另一方。 | ||
|
|
||
| ## 工具 | ||
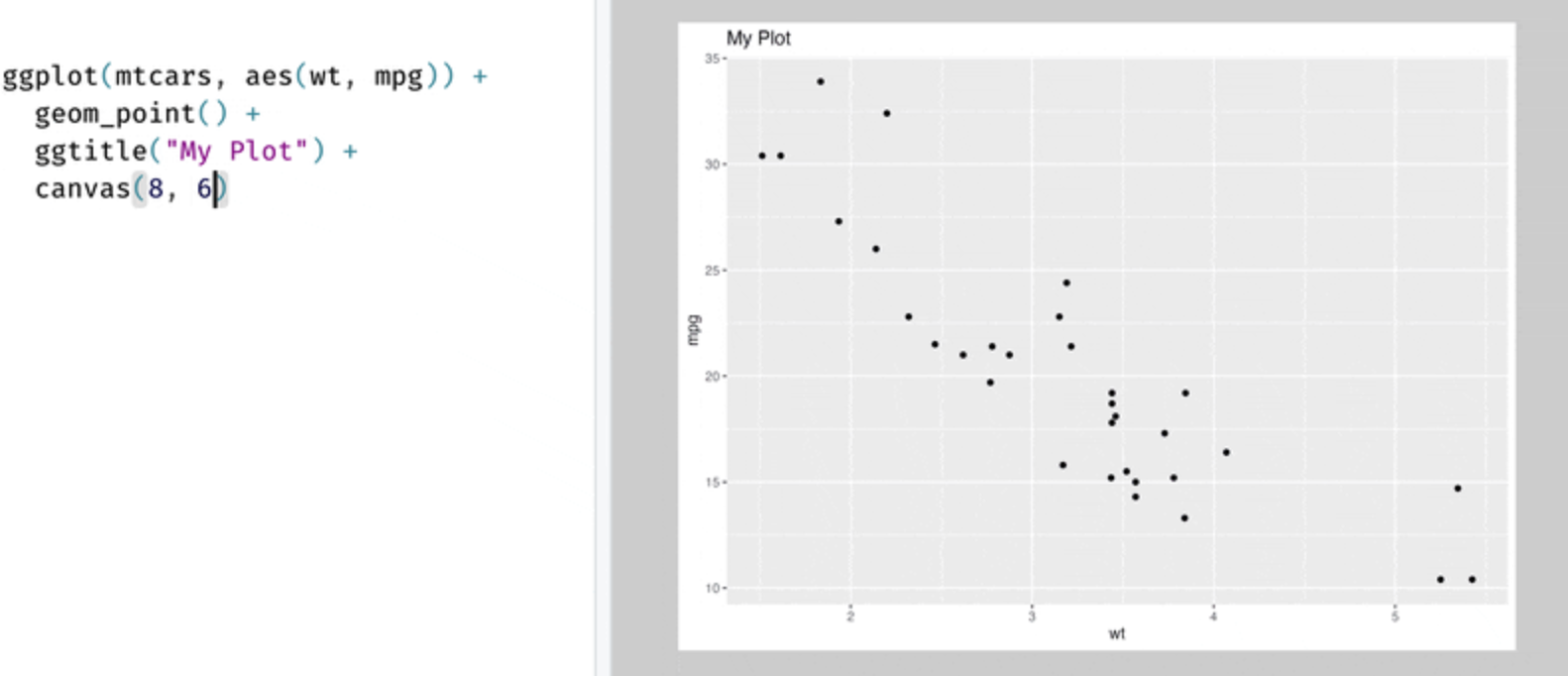
| 8、[ggview | 减少 R 语言图片输出时尺寸设置的试错](https://mp.weixin.qq.com/s/ARTUcjFL0ht0VzB-CMUoMA) | ||
|
|
||
|  | ||
|
|
||
| 使用R语言进行数据可视化时,调整输出图片的尺寸是一个常见的痛点,尤其是需要多次导出图片进行尺寸调整时,不仅耗时,还可能影响工作效率。ggview包通过在RStudio界面中实时预览ggplot2生成的图片尺寸,帮助用户在导出图片前准确调整尺寸,避免了反复试错。 | ||
|
|
||
| - 工具链接:https://github.com/idmn/ggview | ||
|
|
||
|
|
||
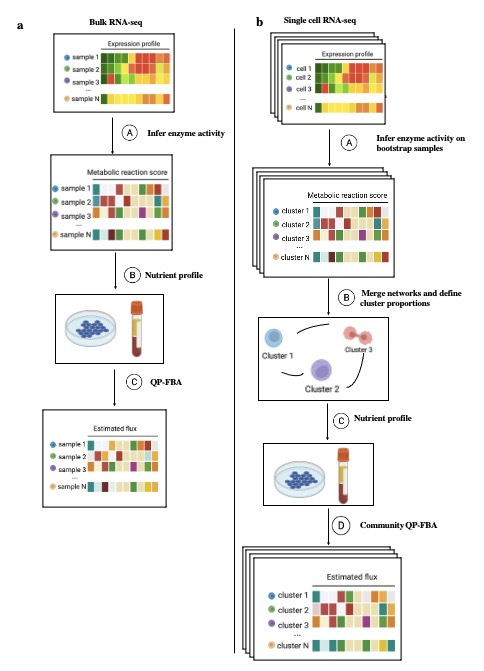
| 9、[METAFlux | 基于R语言的单细胞及Bulk转录组代谢通量分析流程及可视化](https://mp.weixin.qq.com/s/Jg-T_ArrWDZWvP4PuJotDg) | ||
|
|
||
|
|
||
|  | ||
|
|
||
| METAFlux,一款基于R语言的代谢通量分析工具,专门用于从单细胞和Bulk RNA-seq数据中推断代谢通量。该工具于2022年发表在《Nature Communications》上,旨在解决现有代谢组学和通量分析方法在模拟复杂肿瘤微环境时的局限性。METAFlux通过基因组尺度的代谢模型从基因表达数据计算代谢反应活性得分,并应用通量平衡分析来估计代谢通量。它特别适用于肿瘤微环境代谢分析,但也适用于非肿瘤细胞。 | ||
|
|
||
| - 工具链接:https://github.com/milanvarady/Applite | ||
|
|
||
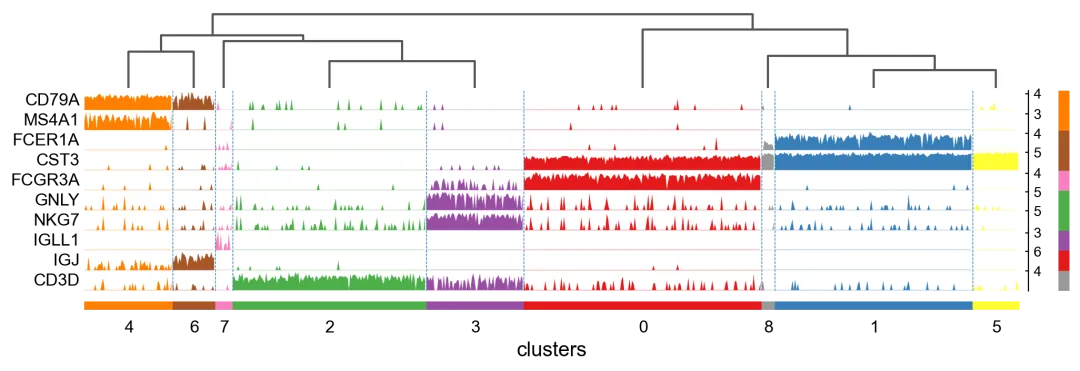
| 10、[tracksPlot | 轨道/热图](https://mp.weixin.qq.com/s/Wja2YdgzOJydVm0GYMdbZQ) | ||
|
|
||
|  | ||
|
|
||
| 本文通过scRNAtoolVis包演示了如何绘制tracksPlot。 | ||
|
|
||
| - 工具链接:https://github.com/junjunlab/scRNAtoolVis | ||
|
|
||
| ## 资源 | ||
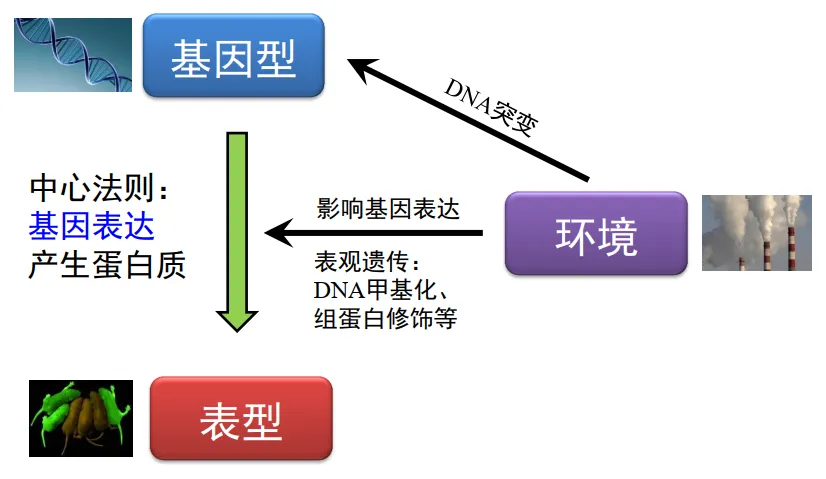
| 11、[可变剪接及其表观遗传调控综述](https://mp.weixin.qq.com/s/rKax5HVLzrySH8p3BfAGiA) | ||
|
|
||
|  | ||
|
|
||
| 本资源根据 2016 年 8 月复旦大学倪挺教授在「表观基因组学暑期国际讲习班」中的报告整理而成。 | ||
|
|
||
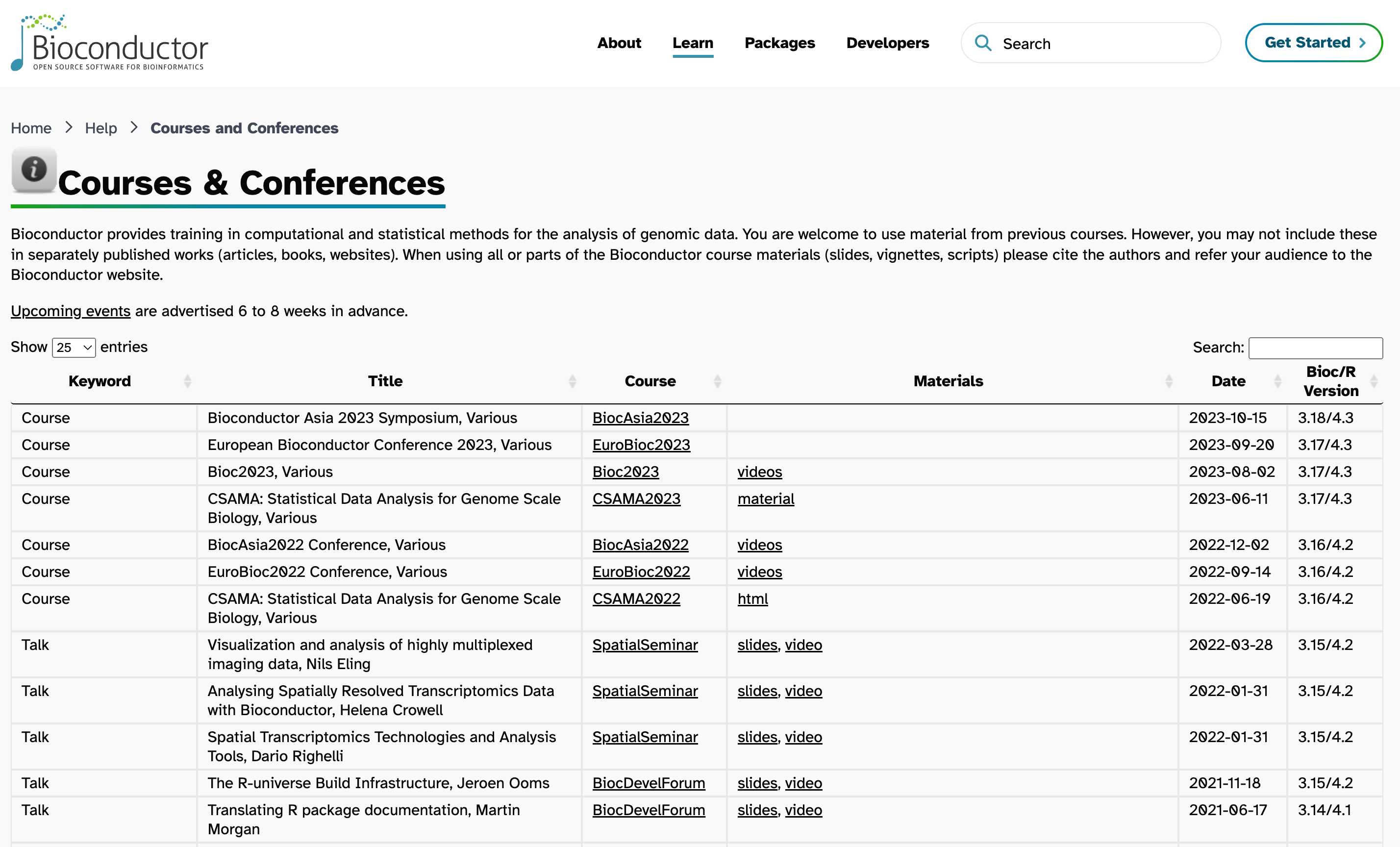
| 12、[Bioconductor既往课程资料集合](https://bioconductor.org/help/course-materials/ "Bioconductor既往课程资料集合") | ||
|
|
||
|  | ||
|
|
||
| 本资源列出了Bioconductor过往基于基因组数据分析的课程材料,并按年份分类。此外,Bioconductor还提供定制化工作坊,针对不同教育和工业客户的需求,提供基因组数据分析的统计方法和软件培训。 | ||
|
|
||
| ## 贡献者(GitHub ID) | ||
|
|
||
| 「Openbiox 生信周刊」运维小队: | ||
|
|
||
| - `@ShixiangWang`(王诗翔) | ||
| - `@kkjtmac`(阚科佳) | ||
| - `@NiEntropy`(赵启祥) | ||
| - `@He-Kai-fly`(何凯) | ||
| - `@JnanZhang`(张佳楠) | ||
| - `@Tomcxf`(陈啸枫) | ||
| - `@wangdepin`(王德品) | ||
| - `@kongjianyang`(空间阳) | ||
| - `@donghongyu2020`(董弘禹) | ||
| - `@DrRobinLuo`(罗鹏) | ||
| - `@Wangcy-rachel` - 王春阳 | ||
| - `@zoe3251` - 舒晨阳 | ||
|
|
||
| ## 订阅 | ||
|
|
||
| 这个周刊每周日发布,同步更新在微信公众号「优雅R」(elegant-r)上。 | ||
|
|
||
| 微信搜索“优雅R”或者扫描二维码,即可订阅。 | ||
|
|
||
|  | ||
|
|
||
|
|
||
| (完) |